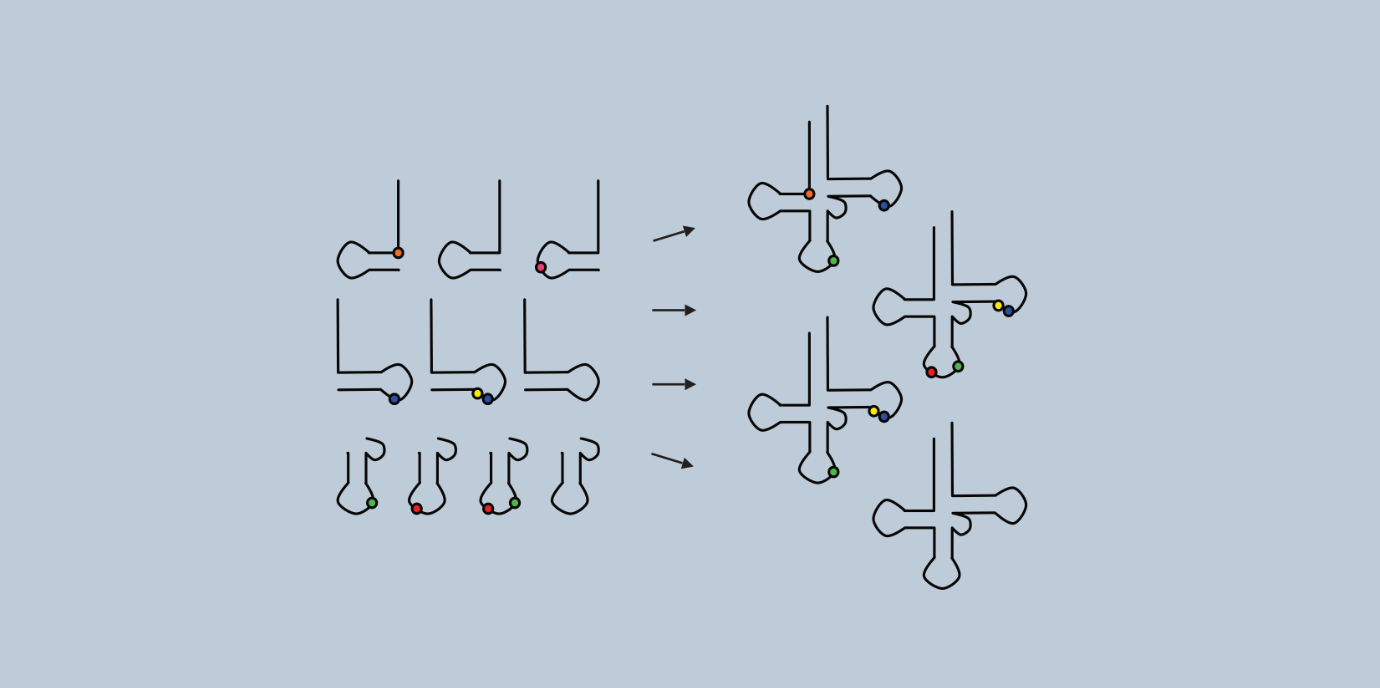Proteinbiosynthese: Selbst gebaute Toolbox gewährt neue Einblicke
Im Fokus des Teams um den Biochemiker Matthias Erlacher stehen die molekularen Wechselwirkungen während der Translation, dem letzten Schritt der Genexpression. Dieser Prozess kann durch Modifikationen von RNA-Nukleotiden in den beteiligten Komponenten der Translation erheblich beeinflusst werden. Mithilfe eines eigens hergestellten Baukastensystems und synthetisch modifizierten tRNAs konnten die ForscherInnen die Rolle einiger dieser Modifikationen charakterisieren.
tRNAs (Transfer-Ribonukleinsäuren) sind für die Übersetzung der genetischen Information in die Aminosäuresequenz eines Proteins unverzichtbar, da sie als Adapter zwischen mRNA (Messenger-RNA) und den entsprechenden Aminosäuren fungieren. Es ist vom genetischen Code vorgegeben, dass Aminosäuren über mehrere verschiedene Codons (synonyme Codons) – eine Abfolge von drei Basen – im genetischen Code repräsentiert werden. Die Codons werden von bestimmten tRNAs erkannt, welche die entsprechende Aminosäure in die Proteinsequenz einfügen. Diese wichtige Funktion ermöglicht es den Zellen, die genetischen Information in Form einer Nukleinsäuresequenz in ein funktionelles Protein zu übersetzen. Das Fundament dafür sind exakt definierte Wechselwirkungen zwischen dem Ribosom (der Proteinfabrik der Zelle), der mRNA (messenger RNA, die Kopie der in DNA gespeicherten genetischen Information) und den tRNAs. Alle an der Proteinsynthese beteiligten RNAs beinhalten modifizierte RNA Bausteine, die die Struktur, Stabilität, Funktion oder den Abbau von RNA-Molekülen modulieren.

BU: Matthias Erlacher und Maria Kompatscher forschen zur Proteinherstellung. (c)Johanna Framm
Erlacher und sein Team haben sich im Detail damit befasst, in welcher Art und Weise RNA-Modifikationen oder Sequenzmotive in tRNAs die Übersetzung des genetischen Codes am Ribosom beeinflussen. Möglich war das mit einem System, das PhD-Studentin Maria Kompatscher am Institut für Genomik und RNomik (Direktor: Alexander Hüttenhofer) – inzwischen hat sie ihr Doktorat erfolgreich abgeschlossen – selbst etabliert hat. Diese Toolbox, bestehend aus chemisch-synthetisch hergestellten RNA Fragmenten, erlaubt das punktgenaue Einfügen von Sequenzen und Modifikationen in RNA Moleküle, welche schließlich in bestimmten Schritten der Translation untersucht werden können. „Dabei können wir sehen, ob eine Modifikation in einer tRNA das Dekodieren von bestimmten mRNA-Codons während der Translation verändert.“ erklärt die Erstautorin der vom FWF geförderten (SFB: RNA deco) und kürzlich im Fachjournal Nucleic Acids Research erschienenen Forschungsarbeit.
Erlacher und Kompatscher haben sich mit Hilfe dieser Toolbox ein ganz besonderes Dekodierungs-Konzept, das sogenannte Superwobbling, angeschaut. „Dieses Konzept des Superwobbelns wird nur von sehr wenigen Organismen oder Zellorganellen verwendet und basiert darauf, dass auf der dritten Codonposition alle vier Basen als korrekt gelesen werden, unabhängig von den ansonsten geltenden Regeln der Basenpaarung (A-U, G-C, G*U)“, erklärt Kompatscher. Die Bakterienart M. mycoides verwendet eine einzelne superwobbling tRNAGly, welche alle vier synonymen Glycin-Codons dekodiert. Im Gegensatz dazu verwenden die meisten Organismen, darunter auch der Modelorganismus E. coli, drei tRNAGly mit unterschiedlichen Anticodons. Zur Überraschung der Forschenden war ein Sequenzelement und waren nicht tRNA Modifikationen oder das Anticodon der tRNA, welches direkt mit der mRNA interagiert, Schlüsselfaktor für Superwobbling. „tRNA Sequenzen und ihre Modifikationen bilden gemeinsam ein komplexes System, welche das tRNA-Molekül darauf trimmen genau und effizient bestimmte Codons während der Translation zu dekodieren“, unterstreicht Erlacher.
Die neue Toolbox, die es erlaubt, aus verschiedenen Fragmenten unterschiedliche tRNAs zu bauen, wollen die ForscherInnen nutzen, um andere supewobbling tRNAs zu charakterisieren und das Dekodieren von modifizierten mRNA-Codons zu modulieren und zu manipulieren.
(9.4.2024, Text: D. Heidegger/red, Bilder: M. Kompatscher (Grafik), J. Framm)
Links:
Contribution of tRNA sequence and modifications to the decoding preferences of E. coli and M. mycoides tRNAGlyUCC for synonymous glycine codons. Maria Kompatscher et al.
Institut für Genomik und RNomik
https://www.i-med.ac.at/rnomics/
FWF Projekt SFB RNA deco
https://www.rna-deco.org/